El M. en C. Ramírez Martínez es estudiante del Doctorado en Ciencias de la Universidad Autónoma del Estado de Morelos (UAEM), y este trabajo es parte de su tesis doctoral.
La Dra. Pastor es profesora-investigadora del Centro de Investigación en Dinámica Celular de la UAEM. Su área de especialidad es la biofísica molecular computacional, con énfasis en el estudio del reconocimiento entre moléculas. Es integrante de la Academia de Ciencias de Morelos.
Esta publicación fue revisada por el comité editorial de la Academia de Ciencias de Morelos.
La formación de grandes complejos macromoleculares, conteniendo muchas proteínas diferentes, es fundamental para que sucedan procesos esenciales para la vida, tales como la replicación y la transcripción del ADN, o la traducción de los ARN mensajeros. Tanto la formación de estos complejos como su correcto funcionamiento están finamente regulados, ya sea por modificaciones en las proteínas y/o en el ADN, o por cambios conformacionales. Ante un panorama que se antoja complicado, llama la atención que los seres vivos contengan proteínas que pueden realizar por sí solas actividades muy sofisticadas sobre el ADN. Tal es el caso de un grupo de proteínas conocidas como recombinasas, las cuales pueden realizar una acción que suena sorprendente: tomar segmentos de ADN y recolocarlos en otro punto del genoma. Sí, tenemos arquitectos que actúan en los genomas.
Uno de estos “arquitectos” es una proteína llamada recombinasa Cre, que actúa sobre una secuencia de ADN conocida como loxP, la cual es identificada por Cre mediante interacciones específicas. Cre es parte del arsenal molecular del bacteriófago P1, un virus que infecta principalmente a la bacteria Escherichia coli, habitante común en nuestros intestinos. Al inicio de la infección a E. coli, Cre desempeña un papel clave al unirse a los sitios loxP localizados en las puntas del cromosoma lineal del fago P1, promoviendo que el cromosoma se convierta en un círculo. Esto permite que el fago P1 pueda vivir dentro de la bacteria sin tener que insertarse en su cromosoma, como hacen otros virus. Por otro lado, cuando E. coli se divide, Cre también se asegura de que a cada célula hija le toque una copia del cromosoma del virus. Esto lo hace nuevamente uniéndose a la secuencia loxP y promoviendo la recombinación de estos sitios, separando así las copias fusionadas del cromosoma de P1, que se forman accidentalmente durante la replicación del genoma viral. Cre es notable porque en este proceso de cortar y unir ADN es muy precisa, no pierde ni inventa información, cosa que sucede con otras maquinarias de recombinación. Otra de las virtudes de Cre es que no requiere la ayuda de nadie ni de fuentes de energía. Son estas características de precisión y autonomía las que la han vuelto una herramienta muy útil en la ingeniería de genomas y una promesa para hacer terapia génica. A pesar de haber sido estudiada intensivamente durante tres décadas, todavía no se acaba de entender cómo identifica a los sitios loxP, ni por qué presenta a veces actividad impredecible en el genoma humano, asunto que la ha mantenido fuera de los ensayos clínicos.
Para que la reacción de recombinación (es decir, el corte y empalme ordenado de dos dúplexes de ADN para generar otros dos dúplexes de ADN) pueda llevarse a cabo, es necesario que se forme un complejo llamado intasoma, compuesto por cuatro proteínas Cre y dos secuencias loxP. Cada secuencia loxP tiene dos sitios en los que se une Cre, dispuestos en espejo uno respecto al otro, y separados por ocho pares de bases. La formación de este complejo sigue una serie ordenada de pasos que comienza con la unión de una proteína Cre a un sitio en loxP. Una vez logrado esto, otra proteína Cre se une al otro extremo de loxP, generando un dímero (Cre2-loxP). Finalmente, dos dímeros se asocian entre sí para formar al intasoma (complejo Cre4-loxP2). El proceso de ensamblado del intasoma se esquematiza en la Figura 1. La proteína Cre está compuesta por los dominios CBD (que contiene a las hélices A a la E) y CAT (que está compuesto por las hélices F a la N y contiene al sitio activo), los cuales son cruciales tanto para la unión a loxP como para promover el corte en el ADN.
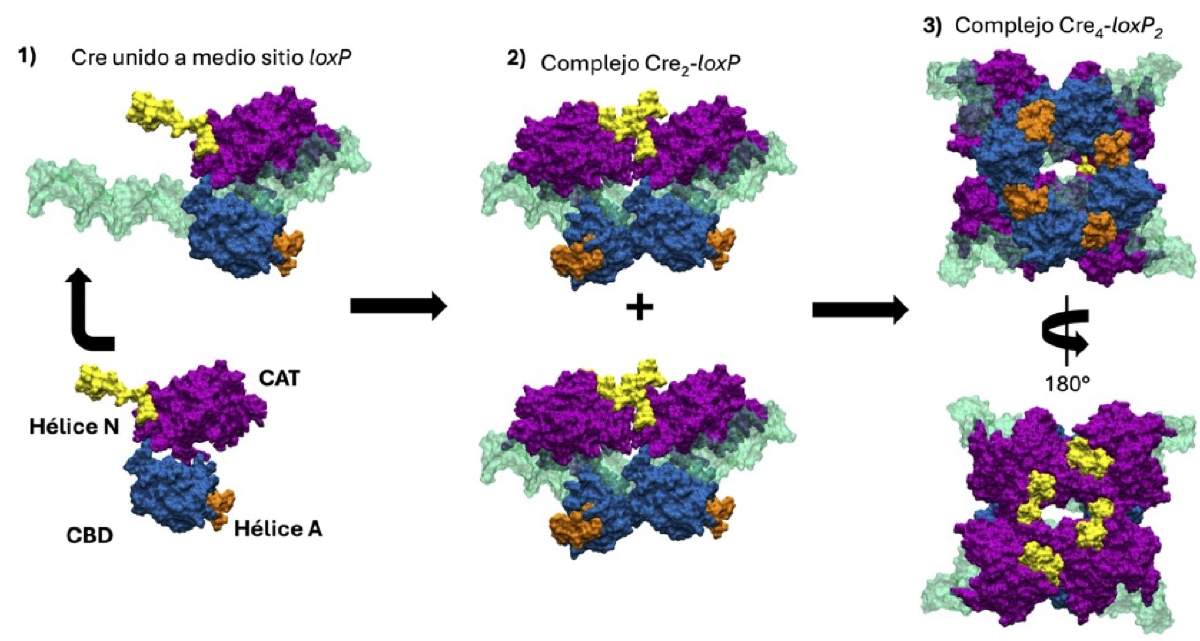
Figura 1. Ensamblado del intasoma. 1) Un monómero de Cre se une a un sitio en loxP (PDB-ID 7RHY), 2) esto permite que otro monómero se una a la otra mitad (PDB-ID 7RHZ); 3) dos dímeros (complejos Cre2-loxP) se asocian para generar un intasoma (el complejo Cre4-loxP2 se muestra en dos orientaciones (PDB-ID 1Q3U)). Cada dominio está marcado en diferente color (CBD, azul; CAT, morado), el ADN (loxP) está en verde. Las hélices A y N, importantes para estabilidad del complejo, están en anaranjado y amarillo, respectivamente. Los códigos PDB-ID se refieren a estructuras accesibles en el Protein Data Bank (https://www.rcsb.org).
El complejo se estabiliza gracias a múltiples interacciones, ya sea entre proteínas vecinas o entre Cre y loxP. Todas estas interacciones son esenciales para evitar que el complejo se deshaga y permitir que la reacción avance. Entre las interacciones más importantes se encuentran las que se establecen entre los dominios CBD, que amarran a Cre de un lado del intasoma (Figura 1, paso 3, lado azul con hélices anaranjadas), y las interacciones entre los dominios CAT, que lo engarzan del otro lado (Figura 1, paso 3, lado morado con hélices amarillas). Sin embargo, aún no entendemos completamente cuáles son los primeros pasos en la formación de este complejo. Además, no sabemos cómo se comporta Cre en ausencia de las interacciones que realiza cuando está en el intasoma, el dímero o unida a un sitio en loxP.
Cre libre es difícil de estudiar experimentalmente
En biología estructural uno de los objetivos es determinar experimentalmente las estructuras de las proteínas, ya sea libres o en complejo con otras moléculas, con el fin de obtener información sobre su función. Sin embargo, es necesario que la proteína cumpla con ciertas características para que esto sea posible.
Supongamos que vamos a un estudio fotográfico para que nos tomen una foto para algún trámite. El fotógrafo amablemente nos pide que no nos movamos para que la foto salga bien, pero decidimos no hacerle caso y empezamos a mover la cabeza de un lado a otro, impidiendo que el fotógrafo logre hacer su trabajo. El resultado de esto, además del regaño del fotógrafo, será una fotografía en la que no se vea nada más que diferentes posiciones de nuestra cabeza, sin poder identificar cada una de sus partes. Sin embargo, si alguien viene y nos detiene la cabeza, entonces sí salimos en la foto, pero también aparecerá la persona que nos detuvo.
Esto mismo ocurre con las proteínas. Algunas se comportan como “piedritas”, casi inmóviles; éstas son fáciles de estudiar, porque su estructura no cambia mucho en el tiempo. Sin embargo, hay otras que sólo pueden ser fotografiadas cuando están agarradas a otra molécula (ya sea otra proteína, ADN, azúcar, etc.), y hay otras que no se pueden fotografiar porque nunca dejan de moverse, aún agarradas a otra molécula, por lo que nunca aparecen en la foto. El caso de Cre corresponde al segundo ejemplo: sólo contamos con información estructural de ella cuando está unida a un sitio en loxP, en un dímero o en el intasoma. Aún no tenemos una estructura de Cre libre, lo cual se podría explicar por un aumento en flexibilidad que presenta cuando no hay una molécula que frene su movimiento; es decir, Cre parece tener una “personalidad dinámica”.
Estudiar la dinámica de Cre libre podría ayudarnos a entender mejor el mecanismo de unión a loxP, así como el papel de cada uno de sus dominios en este proceso. Aunque esto ha sido imposible de hacerse de manera experimental, podemos recurrir a simulaciones computacionales. En el laboratorio de Dinámica de proteínas y ácidos nucleicos de la UAEM utilizamos herramientas computacionales, como las simulaciones de dinámica molecular. Con esta técnica, que se asemeja a un microscopio molecular, podemos observar el movimiento de la proteína y el efecto que tiene la ausencia de las interacciones con otra Cre y/o con loxP en su comportamiento. A continuación, les contamos lo que hemos aprendido a partir de mirar a Cre a través de simulaciones en supercomputadoras como las del Laboratorio Nacional de Supercómputo del Sureste de México en Puebla, el Centro Nacional de Supercómputo en San Luis Potosí, y el Laboratorio Nacional de Cómputo de Alto Desempeño en la Ciudad de México. Estas simulaciones son equivalentes a tener una película de lo que hace Cre; a fin de cuentas, las películas son sucesiones de fotografías ordenadas en el tiempo, y eso es lo que se obtiene de una simulación de dinámica molecular.
Cre es una proteína flexible
Imaginemos el siguiente escenario: un bebé recién nacido, al que su mamá envuelve en una cobija para evitar que mueva los brazos o las piernas. Este bebé se encuentra en un ambiente restrictivo, es decir, los movimientos que puede hacer están limitados por la cobija. Sin embargo, una vez que le quitan la cobija, el bebé vuelve a ser libre de mover los brazos como desee. Ahora, llevemos este ejemplo al nivel molecular. De manera similar, cuando la proteína Cre está integrada en alguno de los complejos (como Cre2-loxP o Cre4-loxP2), establece múltiples interacciones que la mantienen en una conformación fija.
Si eliminamos todas esas interacciones y dejamos que Cre se mueva libremente (como si estuviera dentro de E. coli), podemos observar que la proteína es capaz de estirarse y encogerse, visitando múltiples conformaciones que no se observan cuando está unida a loxP, pero que sí incluyen las que ya se conocen. Esto implica un mecanismo de reconocimiento llamado “selección de conformación”, en el que Cre libre se acomoda de muchas maneras distintas y selecciona una de ellas para unirse a loxP.
La forma más sencilla de clasificar las múltiples maneras en las que se acomoda Cre cuando está libre es calcular su radio de giro. Esto se hace identificando primero el centro de la molécula y luego calculando la distancia promedio de cada átomo de la proteína a ese centro. Esa distancia promedio es el radio de giro. Para entenderlo mejor, pensemos en una manguera con la que regamos las plantas en casa. Cuando la manguera está completamente estirada, su radio de giro es el máximo posible. Sin embargo, cuando la enrollamos sobre un carrete para guardarla, su radio de giro disminuye considerablemente. Este mismo concepto se puede aplicar para interpretar las diferentes conformaciones que puede adoptar la proteína Cre (Figura 2). Separar a Cre de loxP tiene un efecto sobre qué tanto puede extenderse o encogerse la proteína. Esto lo podemos explicar viendo la interacción entre Cre, que tiene carga positiva, con loxP, que tiene carga negativa: al acercarse, la complementariedad de cargas promueve una interacción favorable (cargas del signo opuesto se atraen), pero cuando quitamos la carga negativa de loxP, entonces queda una gran carga positiva en cada dominio de Cre (mostrada en las superficies azules en la Figura 2, particularmente visible en la estructura más compacta), que se repelen entre sí. Esto lleva a que Cre se abra y se cierre, buscando que los dominios interactúen entre sí utilizando diferentes superficies. Cre tiene tantas cargas positivas en su superficie que, en ausencia de ADN, no hay una sola manera de acomodarse que sea realmente favorable. Volviendo a la historia del fotógrafo con la persona que no deja de moverse, ésta es una de las razones por las cuales Cre es notablemente difícil de fotografiar (en términos técnicos, de cristalizarla, o de estudiarla mediante resonancia magnética nuclear o por criomicroscopía electrónica).
Desde el punto de vista funcional, el que Cre libre no se esté quieta y sus dominios se junten y se separen, girando uno respecto al otro todo el tiempo, permite imaginar que cualquiera de los dos dominios podría unirse al ADN y deslizarse sobre él, hasta localizar un sitio loxP; una vez agarrado un dominio, al otro sólo le restaría girar para acomodarse. Este mecanismo de búsqueda se ha propuesto para muchas proteínas que interactúan con ADN, y se considera un método eficiente de búsqueda de sitios específicos porque es una búsqueda en una dimensión (a lo largo del ADN), en lugar de en tres dimensiones.
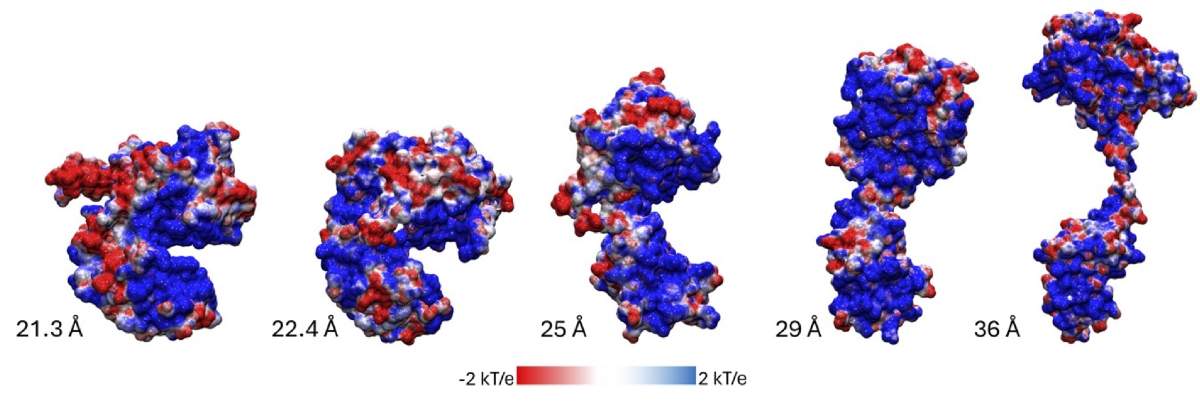
Figura 2. Diferentes conformaciones de Cre con valores crecientes de radio de giro. En todas las estructuras, el dominio CBD se muestra en la parte inferior, así como el valor de radio de giro (medido en Ångström (Å), cada uno equivalente a la diezmilmillonésima parte de un metro). La superficie molecular tiene color rojo si su carga es negativa y color azul si es positiva; cuando es blanca tiene carga neutra.
Los dominios de Cre tienen “bisagras”
Hasta ahora hemos hablado de los movimientos relativos entre los dominios CBD y CAT. Dentro de estos dominios hay hélices conectadas por sectores de la proteína que funcionan como bisagras; estas hélices y su movimiento son fundamentales para establecer las interacciones que mantienen estable al intasoma, amarrando a una Cre con sus vecinas. Además, las interacciones entre Cre determinan si el dominio CAT, que contiene todos los elementos necesarios para realizar el corte en el sitio loxP, se encuentra en una conformación activa o inactiva.
Un elemento clave dentro del dominio CBD es la hélice A, que interactúa con las hélices C y E del dominio CBD vecino (ver en la Figura 1 las hélices marcadas en naranja). Sin embargo, cuando Cre se une al sitio loxP en su forma de dímero o cuando está en su forma monomérica unida a un sitio en loxP, la hélice A adopta una posición diferente de la que tiene en el intasoma. Para entender mejor este comportamiento, podemos imaginar el movimiento de un limpia parabrisas: la varilla de metal se mueve de un lado a otro abarcando toda la superfice del parabrisas. Este movimiento es similar al de la hélice A cuando Cre está libre. Si tomamos al dominio CBD como el parabrisas y a la helice A como la varilla de metal, podemos observar cómo este segmento se mueve de un lado a otro, visitando las conformaciones vistas en el intasoma, el dímero y el monómero unido a un sitio en loxP (vean en la Figura 3 las hélices en verde y rosa mexicano). Para hacer esto más interesante, además de moverse de un lado a otro, la hélice A también puede doblarse y estirarse, adoptando formas similares a un espagueti cocido.
Por otro lado, el dominio CAT tiene una cavidad entre las hélices G-I y las hélices K-L (vean en la Figura 3 las hélices en amarillo y en azul). En esta cavidad se inserta la hélice N de una proteína vecina, lo que permite que se produzcan interacciones entre los dominios CAT, fundamentales para la estabilidad del complejo (vean en la Figura 1 el intasoma con los dominios morados engarzados con hélices amarillas; esas son las hélices N). Si estas interacciones se pierden, la cavidad queda vacía y comienza a mostrar un movimiento de apertura-cierre, similar al de una almeja. Este movimiento es crucial, ya que determina qué tan fácil o difícil es para la cavidad recibir la hélice N vecina, lo cual dependerá de si la cavidad está abierta o cerrada.
Además, la hélice N en sí misma también pierde algunas de sus interacciones y adopta un comportamiento desordenado. Esto significa que pasa de ser un segmento bien estructurado a una forma más flexible, parecida a un espagueti cocido (igual que la hélice A). Esta flexibilidad le permite interactuar con otras partes de Cre, promoviendo la formación de estructuras que ocultan el sitio activo o que imitan las interacciones vistas en el intasoma al interactuar con la cavidad formada por las hélices G, I, K y L (invade su propia cavidad, como si se diera palmadas en la espalda). La interacción con el sitio activo ha sido descrita en experimentos de resonancia magnética nuclear para el dominio CAT aislado, resultando en la propuesta de que Cre vive autoinhibida (imaginen un perro mordiendo su cola) hasta el momento en el que interactúa con el ADN. Lo que vemos en las simulaciones de Cre completa libre es que la hélice N puede hacer muchas cosas además de localizarse en el sitio activo, explicando la rigidez conformacional observada experimentalmente. La Figura 3 resume algunas de estas características de Cre cuando pierde las interacciones típicas del intasoma o del dímero.
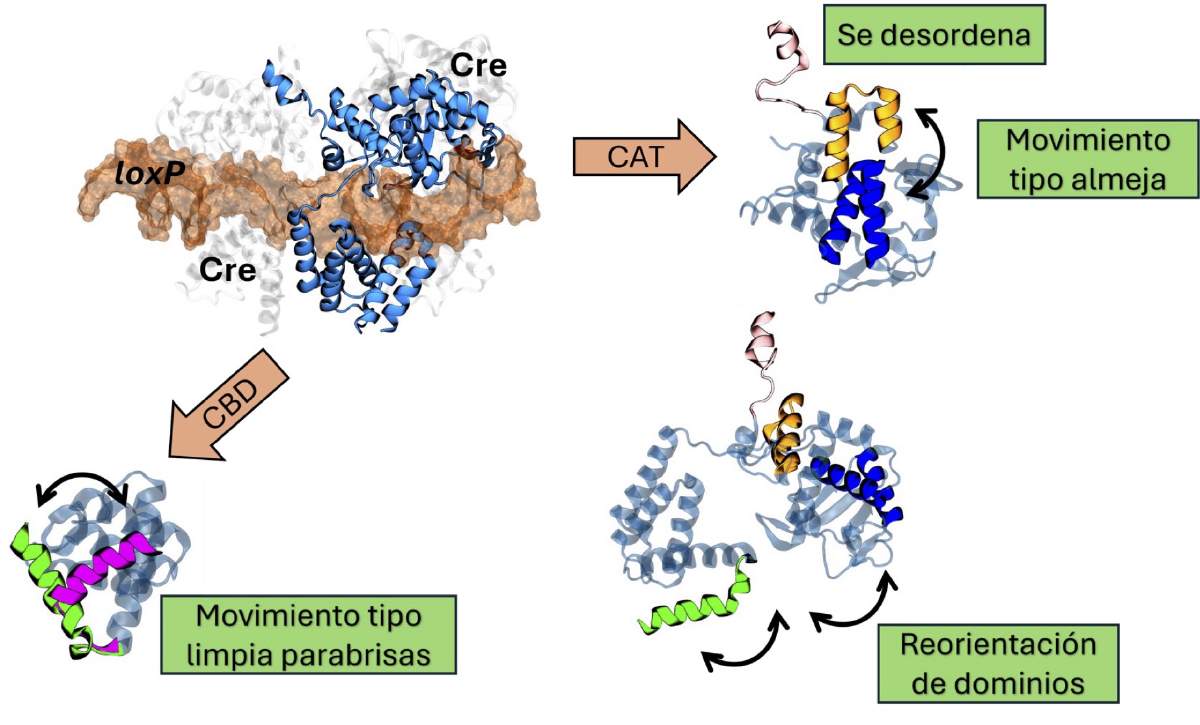
Figura 3. Efecto de la pérdida de interacciones. A la derecha y abajo se muestran todas las regiones descritas y el efecto que tiene el perder interacciones clave en el contexto del intasoma. Se muestran en color las hélices A (rosa mexicano y verde, en las dos conformaciones extremas conocidas experimentalmente), G e I (azul), K y L (amarillo) y N (rosa).
Con lo que les hemos contado, podemos concluir que la proteína Cre, cuando está en complejo con loxP y otras proteínas, adopta una conformación “incómoda”. Es decir, su conformación está siendo limitada por todas las moléculas con las que establece algún tipo de interacción. Cuando estas interacciones se pierden, Cre gana flexibilidad y es capaz de adoptar conformaciones que, aunque podrían no ser funcionales, pueden ser aptas para recibir una secuencia loxP o “cazar” a otra proteína para estabilizarse, iniciando el proceso de formación de un intasoma otra vez. Si quieren leer más sobre Cre, les recomendamos una revisión reciente [1] y dos clásicas [2, 3], donde podrán encontrar notas interesantes sobre su arquitectura, función y aplicaciones.
En este artículo hemos aprendido algo más sobre cómo funcionan estos “arquitectos moleculares”. Aspiramos a comprender, uno de los grandes objetivos de la ciencia, porque como decía Rosalind Franklin, la verdadera descubridora de la estructura del ADN: La belleza de la ciencia reside en su capacidad para desentrañar la complejidad de la naturaleza y revelar su orden subyacente. Comprendiendo este orden estaremos en una mejor posición para emprender modificaciones guiadas por la razón.
Referencias
- Foster MP et al. “Dynamics in Cre-loxP Site-specific Recombination” Current Opinion in Structural Biology 88:102878 (2024)
- Van Duyne GD “Cre Recombinase” Microbiology Spectrum 3:MDNA3-0014-2014 (2015)
- Meinke G et al. “Cre Recombinase and Other Tyrosine Recombinases” Chemical Reviews 116:12785-12820 (2016)
Esta columna se prepara y edita semana con semana, en conjunto con investigadores morelenses convencidos del valor del conocimiento científico para el desarrollo social y económico de Morelos.


