El M. en C. Francisco Murphy estudió la carrera de Bioquímica y Biología Molecular en la entonces Facultad de Ciencias de la UAEM. Durante sus tesis de maestría y doctorado se ha enfocado en buscar estrategias para minimizar el daño por radiación en cristales de proteína.
El Dr. Enrique Rudiño es investigador titular del Instituto de Biotecnología de la Universidad Nacional Autónoma de México (UNAM), uno de los latinoamericanos con más estructuras de proteínas determinadas en su carrera y responsable del grupo de Bioquímica Estructural del Instituto de Biotecnología de la UNAM en Cuernavaca, Morelos. Es integrante de la Academia de Ciencias de Morelos.
Esta publicación fue revisada por el comité editorial de la Academia de Ciencias de Morelos.
Nuestra vida se rige por muchos eventos a nivel molecular, y entre estos, los relacionados con las proteínas son fundamentales. En este contexto, el conocer la forma que tienen estas proteínas es una aventura que miles de científicos de todo el mundo, incluyendo a los de Morelos, han abordado en los últimos 50 años; sin embargo, como en cualquier otra actividad humana, es posible encontrar errores en esas estructuras. Estos errores tienen su origen tanto en las técnicas utilizadas para descubrir la estructura tridimensional de las proteínas, así como en cuestiones mucho más humanas relacionadas con la ética y la integridad científica. Para entender estas relaciones y su importancia los invitamos a leer esta historia.
Ciencia y proteínas
Uno de los grandes objetivos de la humanidad, desde hace cientos o miles de años, ha sido comprender cómo funcionamos. Una manera de acercarnos a esta meta es a través de la bioquímica, la disciplina que estudia la química de los organismos vivos. Muchos estudios bioquímicos se enfocan en las proteínas, ya que desempeñan un papel esencial en la vida tal como la conocemos. La comprensión de qué son y cómo funcionan las proteínas ha transformado nuestra forma de ver la vida y ha impulsado avances que han cambiado el mundo. Pero, ¿sabe usted qué son exactamente las proteínas? En casi cualquier enciclopedia se puede encontrar una definición similar a la siguiente: las proteínas son macromoléculas formadas por cadenas lineales de aminoácidos. El prefijo macro proviene del griego y significa grande, lo que indica que las proteínas son moléculas grandes, compuestas por aminoácidos. Sin embargo, sin tener una idea clara de qué son las moléculas o los aminoácidos, esta definición puede resultar poco útil. A continuación, se ampliará esta definición para que usted, querido lector, no solo comprenda qué son las proteínas, sino que también las aprecie como las maravillas que son. Antes de continuar, es importante aclarar algunos conceptos esenciales.
Átomos y moléculas
Los átomos están compuestos por tres partículas subatómicas: protones, neutrones y electrones. Los protones y los neutrones forman el núcleo del átomo, mientras que los electrones orbitan alrededor. Una propiedad de estas partículas es su carga eléctrica: los electrones tienen carga negativa, los protones poseen carga positiva y los neutrones no tienen carga (son neutros). En los organismos vivos, los átomos más comunes son el carbono, el hidrógeno, el oxígeno, el nitrógeno, el fósforo y el azufre (C, H, O, N, P y S, por sus respectivos símbolos químicos en la famosa Tabla Periódica). En la naturaleza, la mayoría de los átomos se unen para formar moléculas o compuestos. La unión entre dos átomos implica que cada uno comparte al menos un electrón, lo que define un enlace químico. En este sentido, el carbono puede formar hasta cuatro enlaces, el nitrógeno tres, el oxígeno dos y el hidrógeno uno. Por ejemplo, en el metano (CH4), el carbono comparte un electrón con cada uno de los cuatro átomos de hidrógeno, que a su vez comparten su único electrón con el carbono, formando una molécula con cuatro enlaces. Algo similar sucede en el amoniaco (NH3), el agua (H2O) y el hidrógeno molecular (H2) que forman moléculas con tres, dos y un enlace, respectivamente.
En nuestro planeta existen todo tipo de moléculas, desde las más simples hasta las más complejas. Según su complejidad, una molécula puede estar formada por varios átomos distintos. Ciertos grupos de átomos, llamados grupos funcionales, influyen en el comportamiento de la molécula y, por ende, en sus propiedades físicas. Por ejemplo, comparemos la solubilidad del etano (C2H6) y del etanol (C2H6O) en agua. El etano está compuesto por dos átomos de carbono unidos entre sí, y cada uno está unido a tres átomos de hidrógeno. En el caso del etanol, uno de estos hidrógenos se sustituye por un grupo funcional hidroxilo (-OH), añadiendo un átomo de oxígeno a la molécula (Figura 1). El etanol se disuelve fácilmente en agua, pero el etano no. En el caso del etanol, el oxígeno del grupo hidroxilo hace que la molécula sea polar, es decir, que sus electrones están distribuidos de manera desigual en la molécula, creando un extremo más positivo y otro más negativo (igual que un imán). Esta polaridad permite al etanol interactuar con otras moléculas polares, como el agua, lo que explica su capacidad para disolverse fácilmente en este solvente. Este ejemplo ilustra cómo un pequeño cambio en una molécula puede modificar drásticamente sus propiedades.
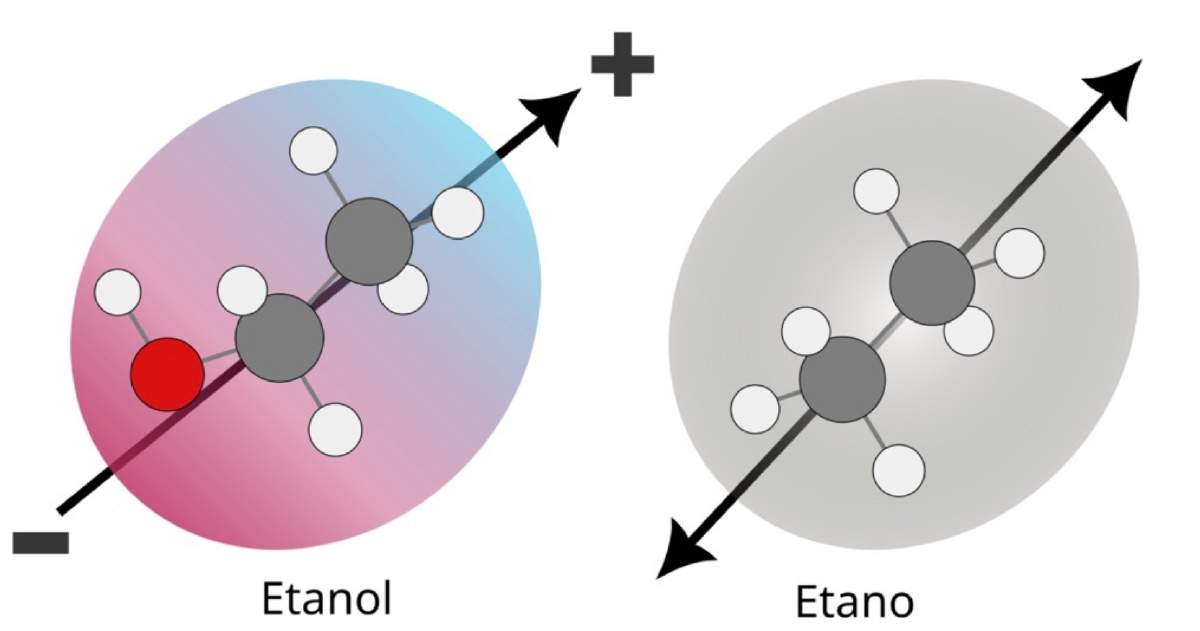
Figura 1. Comparación entre etanol y etano— Los átomos se representan como esferas y los enlaces como líneas. El código de colores es el siguiente: rojo para el oxígeno, gris para el carbono y blanco para el hidrógeno. El oxígeno es un átomo muy electronegativo, lo que significa que atrae electrones hacia sí mismo. Esto provoca que la región de la molécula cercana al oxígeno sea rica en electrones, mientras que el otro extremo es pobre en ellos. Como resultado, la molécula de etanol tiene cierta polaridad, representada con un gradiente de colores: de azul (positivo) a rojo (negativo). No sucede esto en el etano, porque la distribución de electrones en la molécula es igual en ambos extremos.
Aminoácidos y proteínas
Una vez comprendido qué son las moléculas y los grupos funcionales, podemos ahora definir los aminoácidos y, más adelante, mejorar la definición de las proteínas mencionada en la introducción. Un aminoácido es una molécula compuesta por un átomo de carbono al que están unidos: un grupo amino (-NH2), un grupo carboxilo (-COOH), un átomo de hidrógeno (-H) y una cadena lateral (-R). Esta última varía en función del aminoácido en cuestión y puede ir desde un simple átomo de hidrógeno, hasta un grupo funcional más complejo.
El interior de una célula está repleto de moléculas: agua, azúcares, aminoácidos, proteínas, ácidos nucleicos, etc. Es en este entorno donde tiene lugar la formación de proteínas. Durante este proceso, los aminoácidos se van uniendo uno a uno de manera ordenada, lo que explica el término cadenas lineales en la definición enciclopédica. Aunque existen muchos aminoácidos, únicamente veinte participan en la formación de la mayoría de las proteínas que conocemos. Cabe destacar que, durante su unión, los aminoácidos experimentan ligeras modificaciones. Por este motivo, los aminoácidos que forman parte de una proteína se denominan técnicamente como residuos de aminoácidos o simplemente residuos. Las proteínas son tan importantes para la vida que constituyen más de la mitad del peso seco de una célula. Esto tiene sentido si consideramos la gran variedad de tareas que realizan. En general, cada proteína tiene una función específica, pero, ¿cómo funcionan las proteínas?
Estructura tridimensional de proteínas
En resumen, la polaridad influye en la forma en que una molécula interactúa con otras. Cuando los electrones se distribuyen de manera uniforme, como en el etano, la molécula es no polar; pero si la distribución es desigual, como en el etanol, la molécula es polar. Además, algunas moléculas pueden tener una carga completa, positiva o negativa, debido a que en algún momento de su existencia han perdido o ganado un electrón. De manera similar, los aminoácidos se pueden clasificar, según su cadena lateral, en polares y no polares, y algunos pueden tener carga positiva o negativa.
En el ambiente acuoso (polar) dentro de la célula, los residuos de una proteína en formación, buscan un entorno favorable según sus características fisicoquímicas, entre las cuales la polaridad, por ejemplo, es importante. Esto provoca que la proteína se doble, o se pliegue, sobre sí misma. El plegamiento de las proteínas hace que cada una adopte una estructura tridimensional única. En resumen, las proteínas son una cadena de aminoácidos unidos que forman una molécula gigante con la estructura adecuada que le permite realizar su función. En esencia, las proteínas son máquinas moleculares, evolucionadas a lo largo de miles de millones de años. La biología estructural es la ciencia que estudia cómo la estructura de las moléculas biológicas determina su función. Como podrá imaginar, las proteínas son el objeto de estudio de gran parte de los biólogos estructurales. Cabe señalar que también existen proteínas que, debido a su función, no tienen una estructura tridimensional fija.
El Banco de Datos de Proteínas (PDB)
Hasta hace poco, la única forma de obtener la estructura tridimensional de una proteína era mediante experimentos. Sin embargo, en 2021 se publicó un artículo que sorprendió a la comunidad científica. En él se demostró que era posible predecir la estructura de las proteínas mediante un sistema de inteligencia artificial conocido como alphafold2, algo que hace una década parecía imposible. Gracias a este avance, Demis Hassabis y John M. Jumper, autores del artículo, recibieron la mitad del Premio Nobel de Química de este año, en reconocimiento a su innovadora metodología.
La certeza de estas predicciones es en lo general muy alta pero depende de diversos factores, por ejemplo, la presencia de proteínas similares en nuestras bases de datos. Si alphafold2 nunca se encontró en su entrenamiento algo parecido, la predicción de la estructura de la proteína de interés será imprecisa. Por tanto, a veces, es preferible realizar un experimento a confiar ciegamente en una predicción. De cualquier manera, a través de predicciones o mediante experimentos, el resultado final es un modelo atómico: un archivo de texto con las coordenadas en tres dimensiones de los átomos que conforman la proteína y un valor de la confianza que podemos darles a estas coordenadas. Estos modelos se depositan en el Banco de Datos de Proteínas (PDB, por sus siglas en inglés), el repositorio más relevante en biología estructural (https://www.rcsb.org/).
Sin embargo, como dice el dicho, no todo lo que brilla es oro. Dejando de lado los modelos obtenidos por medio de predicciones, a veces sucede que los modelos experimentales contienen errores. En ciertas ocasiones, las prisas o nuestra falible humanidad provocan ligeros errores que pasan desapercibidos, incluso para el experto más versado en la materia (Figura 2). También, en raras ocasiones, quizá por la presión académica por publicar artículos se han depositado modelos fraudulentos: intentos deliberados de engañar a la comunidad científica. Tarde o temprano, estos fraudes son descubiertos y acaban con la carrera científica de los culpables.
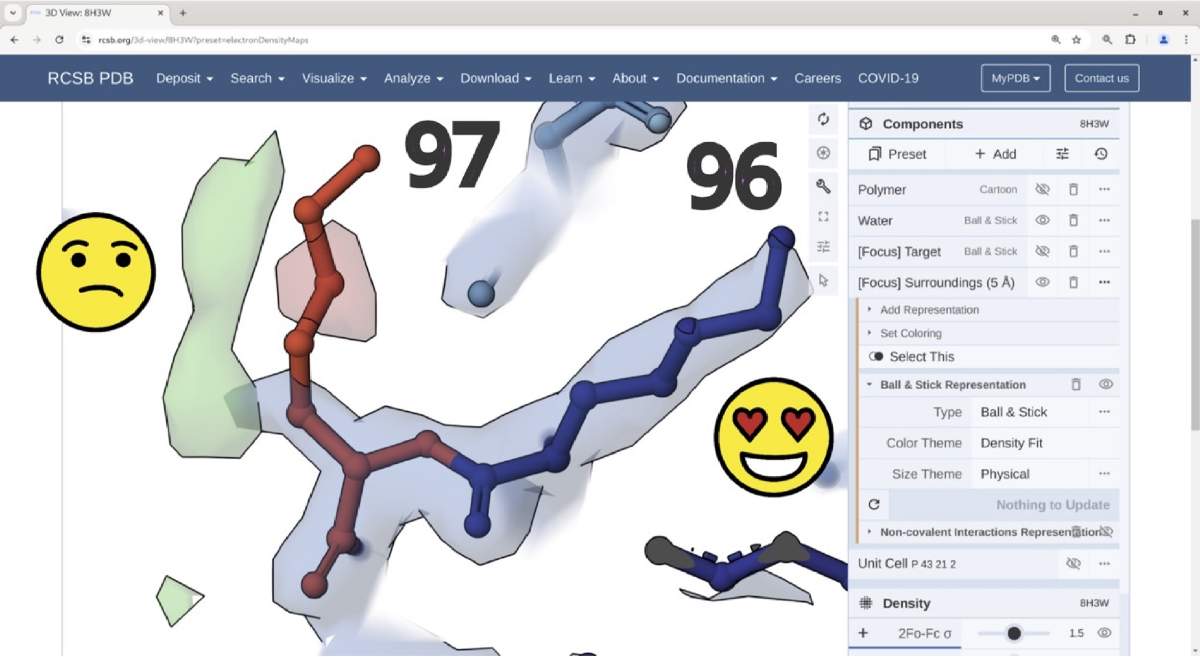
Figura 2. Error humano— Se observan dos residuos de un modelo atómico depositado en el PDB (8H3W). Estos modelos se construyen utilizando la densidad electrónica (las nubes azules), como guía. Cuando el modelo no sigue correctamente esta referencia, aparecen nubes adicionales en verde y rojo. Lo que implica que al modelo atómico le faltan (nube verde) o le sobran (nube roja) átomos en ese lugar. En este caso, los residuos se han coloreado según qué tan bien coinciden con la densidad electrónica. El residuo 96 está correctamente modelado, mientras que el 97 debería estar donde aparece la nube verde.
Daño por radiación
El método más común para obtener la estructura de una proteína es la difracción de rayos X por cristales. En resumen, este experimento consiste en cristalizar una proteína de interés y, mientras el cristal rota, exponerlo a un haz de rayos X. La rotación se realiza en pasos discretos, del orden de fracciones de grado, y la rotación total suele ser bastante amplia, a este proceso se le conoce como colecta de datos. Como resultado, el cristal se expone muchas veces a dichos rayos. Las consecuencias aparecen casi de inmediato, la radiación utilizada tiene la energía suficiente para ionizar las moléculas que componen el cristal, es decir, arrancar electrones de sus átomos. Esto provoca la generación de radicales libres, especies altamente reactivas capaces de interactuar con cualquier molécula a su paso, incluida la proteína. Esta interacción no deseada modifica la estructura de la proteína cristalizada. De hecho, es común que algunos modelos depositados en el PDB presenten modificaciones estructurales debido a este fenómeno. Algunos enlaces químicos pueden romperse; especialmente los que se forman entre átomos de azufre, conocidos como puentes disulfuro (Figura 3). Literalmente, estas modificaciones no son errores en el modelo, sino una representación precisa de lo que ocurre en el cristal. No obstante, dado que los modelos atómicos se utilizan para generar hipótesis, como en el diseño de fármacos, es crucial comprender hasta qué punto y de qué manera la estructura ha sido alterada. De lo contrario, una mala interpretación de la estructura podría llevar a hipótesis erróneas e invalidar todo el trabajo posterior.
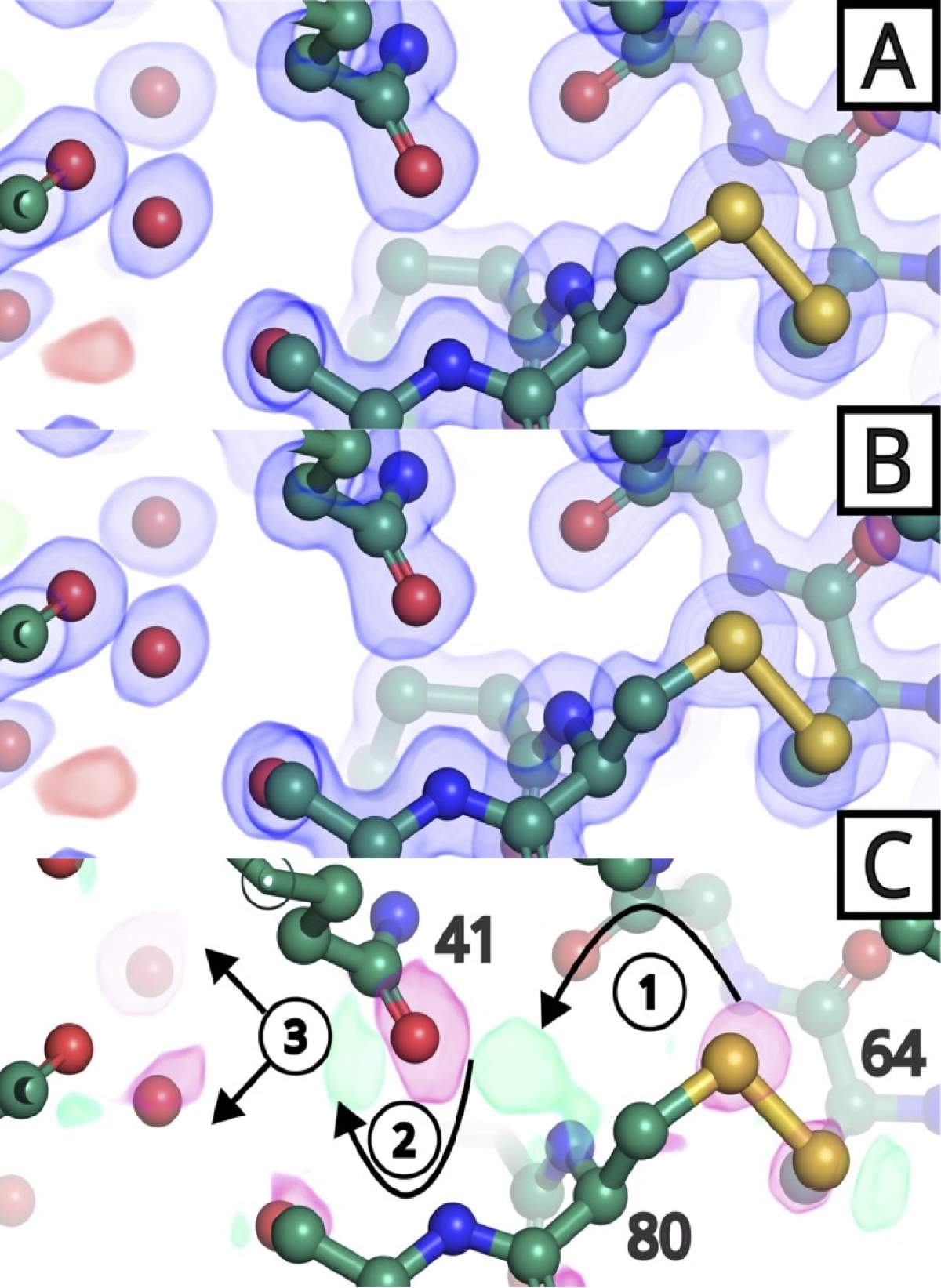
Figura 3. Ruptura de un puente disulfuro— Código de colores: rojo para el oxígeno, verde para el carbono, amarillo para el azufre y azul para el nitrógeno (los hidrógenos no suelen verse en estos modelos). En [A], se muestran los resultados de una primera colecta de datos, el modelo atómico y su densidad electrónica (nubes en color azul). En [B], igual que en [A], pero este es el producto de una segunda colecta de datos sobre el mismo cristal, con mayor exposición a los rayos X. En [C], se ilustra cómo se ve el daño por radiación, restando la densidad electrónica entre colectas. Ahora las nubes en cian y magenta son similares a las nubes verdes y rojas de la figura anterior. La distinción es que indican cambios ocurridos entre las colectas. Los átomos de azufre del puente disulfuro, formado por los residuos 64-80, tienen menor densidad electrónica en la segunda colecta de datos, lo que sugiere su ruptura. El residuo 80 adopta una nueva posición (1), provocando un reajuste en el residuo 41 (2), y desplazando dos moléculas de agua cercanas (3).
Estudiar la estructura tridimensional de las proteínas es esencial, pero también lo es comprender cómo se obtiene esta información para evitar errores de interpretación, especialmente para quienes se adentran en el fascinante mundo de la biología estructural, o bien, quieren utilizar indirectamente el acervo experimental de estructuras de proteínas mediante el uso de herramientas de inteligencia artificial. En Cuernavaca, Morelos, se encuentra el laboratorio de Bioquímica Estructural del Instituto de Biotecnología de la UNAM, donde, entre otros temas, investigamos los efectos de la radiación en diferentes proteínas. Si está interesado en aprender más sobre este tema, lo invitamos a enviarnos un correo electrónico o a visitar nuestro laboratorio.
Para aprender más
Rudiño Piñera, Enrique. ¿Las proteínas tienen una forma tridimensional? y eso ¿cómo afecta mi vida? La Unión de Morelos, 21 de noviembre de 2023. (https://acmor.org/publicaciones/las-prote-nas-tienen-una-forma-tridimensional-y-eso-c-mo-afecta-mi-vida)
Pastor Colón, Nina y Enrique Rudiño Piñera. Origami molecular y el premio Nobel de química 2024. La Unión de Morelos,21 de octubre de 2024. (https://acmor.org/publicaciones/origami-molecular-y-el-premio-nobel-de-qu-mica-2024)
Esta columna se prepara y edita semana con semana, en conjunto con investigadores morelenses convencidos del valor del conocimiento científico para el desarrollo social y económico de Morelos.


